연구팀은 인체의 27개 부위 조직의 3차원 게놈 지도를 분석해 치매, 심혈관계 질환 등을 포함한 2만 7천여 개 이상의 복합 질환 관련 유전 변이 기능을 예측했다.

정인경 교수(좌), 이정운 박사과정(우).[사진=카이스트 제공]
정인경 교수, 빙 렌 교수가 공동 교신 저자로 참여한 이번 연구 결과는 국제 학술지 ‘네이처 제네틱스(Nature Genetics) 9월 10일 자 온라인판에 게재됐다. (논문명 : A compendium of promoter-centered long-range chromatin interactions in the human genome)
현재까지 수많은 연구를 통해 알츠하이머병, 파킨슨병, 자가면역질환 등 다양한 복합 질환의 원인을 규명하려는 시도가 이뤄지면서 실제 다수의 질환과 관련한 중요 유전변이가 발견됐다.
이에 지난 10년간 비약적으로 발전한 3차원 게놈 구조 연구는 비전사 지역에 존재하는 유전변이도 3차원 게놈 구조에 의해 형성되는 염색질 고리 구조(chromatin loop)를 통해 멀리 떨어진 유전자를 조절할 수 있다는 모델을 제시했다.
그러나 이러한 3차원 게놈 구조 연구는 몇 가지 세포주를 대상으로만 국한돼 있고, 질환과 직접 연관이 있는 각 인체 조직을 표적으로 한 게놈 3차 구조는 규명되지 않았다.
연구팀은 인체 내의 27개 조직을 대상으로 이들 게놈의 3차원 구조를 규명하기 위해 전사촉진 부위만 선택적으로 분석하는 ‘표적 염색질 3차 구조 포착법(promoter-capture Hi-C)’이라 불리는 신규 실험 기법을 활용해 고해상도의 3차원 게놈 참조 지도를 작성했다.
그 결과 인간 게놈에 존재하는 약 90만 개의 게놈 3차원 염색질 고리 구조를 발굴하고, 이들 중 상당수가 각 인체 조직 특이적으로 존재한다는 사실도 규명했다.
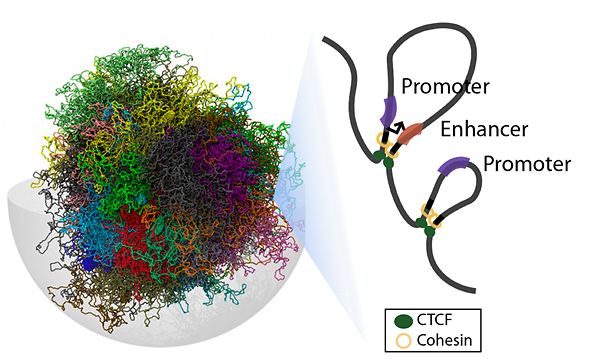
3차원 게놈 구조 모식도.[사진=카이스트 제공]
연구팀은 3차원 게놈 구조를 기반으로 지금까지 기능이 명확하게 정의되지 않은 2만 7천여 개 이상의 질환 연관 유전 변이의 표적 유전자를 정의해 이들 변이의 기능을 예측했다. 나아가 각 질환의 표적 유전자 유사도에 기반해 질환과 질환 사이의 신규 관계를 규명했고, 이를 바탕으로 여러 질환에 공통으로 관여하는 신규 분자 기전을 제시했다.
정 교수는 “복합 질환 기전 규명을 위해 비전사 게놈의 중요성을 강조하고 존재하는 다수의 중요 유전변이를 3차원 게놈 구조 해독을 통해 규명 가능함을 보였다”라며 “이번 결과는 퇴행성 뇌 질환을 포함 다양한 복합 질환의 신규 기전 규명 및 표적 발굴에 활용될 것이다”라고 말했다.
이번 연구는 한국연구재단 신진연구자지원사업, 보건복지부 질환극복기술개발사업, 서경배 과학재단의 지원을 통해 수행됐다.
©'5개국어 글로벌 경제신문' 아주경제. 무단전재·재배포 금지



![[르포] 중력 6배에 짓눌려 기절 직전…전투기 조종사 비행환경 적응훈련(영상)](https://image.ajunews.com/content/image/2024/02/29/20240229181518601151_258_161.jpg)



